Nouvelle stratégie pour affiner l’édition CRISPR
Vous voulez garder la racaille hors de la fête du pool génétique ? Faufilez-vous et claquez la porte avant qu’ils n’arrivent.
C’est l’idée centrale d’une nouvelle stratégie des scientifiques de l’Université Rice qui cherchent à éviter les erreurs d’édition de gènes en ajustant à l’avance des stratégies d’édition spécifiques basées sur CRISPR.
L’ingénieur chimiste et biomoléculaire du riz Xue Sherry Gao et le chimiste Anatoly Kolomeisky et leurs laboratoires ont combiné théorie et expérimentation pour une approche globale visant à créer de meilleurs éditeurs de base, des machines moléculaires qui ciblent et réparent l’ADN défectueux à une résolution à base unique.
Leur travail apparaît dans Nature Communications.
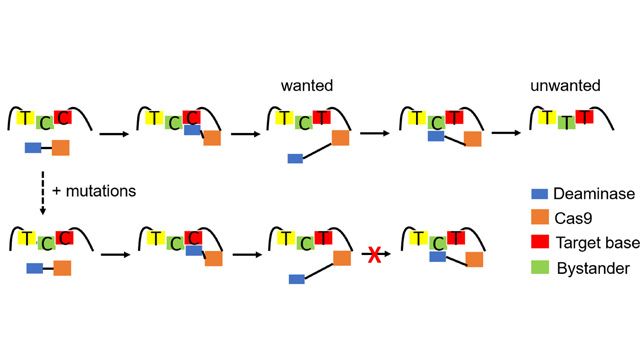
L’article décrit les processus moléculaires que les éditeurs de bases utilisent pour manipuler les brins d’ADN, les coupant si nécessaire et faisant place au code de remplacement. Quand cela fonctionne, comme il le fait de plus en plus pour traiter des maladies génétiques comme la drépanocytose et certains cancers, l’éditeur ne modifie que le nucléotide prévu.
Et quand ce n’est pas le cas, c’est parce que les modifications apportées par les spectateurs peuvent provoquer des effets indésirables.
La stratégie Rice cherche principalement à éliminer les modifications capricieuses des spectateurs, les nucléotides adjacents à la cible de l’éditeur de base. Le laboratoire de Gao a précédemment introduit des outils pour améliorer la précision des modifications basées sur CRISPR des mutations de la cytosine jusqu’à 6 000 fois.
Pour le nouveau projet, elle a engagé le laboratoire Kolomeisky pour aider à créer un cadre théorique pour éliminer les essais et les erreurs dans la conception d’une bibliothèque d’éditeurs. Ceux-ci cibleraient mieux les mutations qui causent la maladie tout en évitant les passants. Dans le processus, le cadre pourrait aider les scientifiques à mieux comprendre les processus chimiques et physiques qui se déroulent lors de l’édition de la base.
« Sherry et d’autres scientifiques expérimentaux avaient déjà des résultats qui fonctionnaient », a déclaré Kolomeisky, se référant à l’article précédent, dans lequel le laboratoire utilisait son éditeur pour convertir les cytosines en thymines, corrigeant les mutations de l’ADN tout en évitant les cytosines autrement vulnérables en amont. « Mais malgré ces développements étonnants, il n’y a eu aucune compréhension microscopique de ce que nous devons faire avec ces systèmes de protéines pour améliorer l’édition. »
Il a déclaré que Qian Wang, ancien chercheur postdoctoral dans le laboratoire de Kolomeisky et maintenant professeur adjoint à l’Université des sciences et technologies de Chine (USTC), Hefei, avait relevé le défi en utilisant l’expérience de la cytosine de Gao comme référence.
« Nous avons appliqué le modèle pour ce résultat et obtenu quelques paramètres importants que nous avons ensuite utilisés pour concevoir quelles mutations et où sont nécessaires pour obtenir une édition précise », a déclaré Kolomeisky. « En fin de compte, cette symbiose de la théorie et de l’expérimentation nous permet de travailler de manière intelligente. »
Leur stratégie combine des simulations de dynamique moléculaire et des modèles stochastiques (alias aléatoires) qui identifient les énergies de liaison entre les molécules nécessaires pour atteindre une sélectivité d’édition maximale. Des expériences dans le laboratoire de Gao ont validé les résultats.
De manière critique, le cadre comprend un moyen de caractériser l’affinité de liaison entre les désaminases – des enzymes qui catalysent l’élimination d’un groupe aminé d’une molécule – et l’ADN simple brin (ADNsb).
Idéalement, ont-ils dit, la désaminase reste sur l’ADNss juste assez longtemps pour terminer l’édition principale et se libère avant d’éditer par inadvertance un site témoin.
« La chose importante ici est qu’une mutation ne fonctionne pas pour différents systèmes », a déclaré Kolomeisky. « Donc, pour chaque système, vous devez refaire cette procédure, mais au moins ce qui doit être fait est clair. »
« Le modèle a très bien réussi à refléter ce qui a déjà été fait expérimentalement », a déclaré Gao. « Mais depuis lors, nous avons été en mesure de refuser les effets de spectateur dans d’autres systèmes d’édition de base.
« Parce que le nombre de mutants pourrait se chiffrer en milliers, il est irréaliste pour les expérimentateurs seuls de vérifier les éditeurs de base individuels », a-t-elle déclaré. « Seule cette approche multidisciplinaire nous permettra de créer une énorme bibliothèque d’éditeurs par ordinateur, puis de réduire les nombres aux candidats les plus prometteurs pour des vérifications expérimentales supplémentaires. C’est ce vers quoi nous travaillons.
Référence: Wang Q, Yang J, Zhong Z, Vanegas JA, Gao X, Kolomeisky AB. Un cadre théorique général pour concevoir des éditeurs de base avec des effets de spectateur réduits. Nat Commun. 2021;12(1):6529. doi: 10.1038/s41467-021-26789-5
Cet article a été republié à partir de ce qui suit matériaux. Remarque : le matériel peut avoir été modifié pour sa longueur et son contenu. Pour plus d’informations, veuillez contacter la source citée.